-
Notifications
You must be signed in to change notification settings - Fork 2
/
08_datentransformation.Rmd
376 lines (280 loc) · 20.2 KB
/
08_datentransformation.Rmd
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
98
99
100
101
102
103
104
105
106
107
108
109
110
111
112
113
114
115
116
117
118
119
120
121
122
123
124
125
126
127
128
129
130
131
132
133
134
135
136
137
138
139
140
141
142
143
144
145
146
147
148
149
150
151
152
153
154
155
156
157
158
159
160
161
162
163
164
165
166
167
168
169
170
171
172
173
174
175
176
177
178
179
180
181
182
183
184
185
186
187
188
189
190
191
192
193
194
195
196
197
198
199
200
201
202
203
204
205
206
207
208
209
210
211
212
213
214
215
216
217
218
219
220
221
222
223
224
225
226
227
228
229
230
231
232
233
234
235
236
237
238
239
240
241
242
243
244
245
246
247
248
249
250
251
252
253
254
255
256
257
258
259
260
261
262
263
264
265
266
267
268
269
270
271
272
273
274
275
276
277
278
279
280
281
282
283
284
285
286
287
288
289
290
291
292
293
294
295
296
297
298
299
300
301
302
303
304
305
306
307
308
309
310
311
312
313
314
315
316
317
318
319
320
321
322
323
324
325
326
327
328
329
330
331
332
333
334
335
336
337
338
339
340
341
342
343
344
345
346
347
348
349
350
351
352
353
354
355
356
357
358
359
360
361
362
363
364
365
366
367
368
369
370
371
372
373
374
375
376
---
title: "Datentransformation Advanced"
author: CorrelAid e.V.
date: "`r Sys.Date()`"
authors:
- Zoé Wolter
- Jonas Lorenz
output:
html_document:
toc: true
toc_depth: 2
toc_float: true
theme: flatly
css: www/style.css
includes:
after_body: ./www/favicon.html
language: de
runtime: shiny_prerendered
---
```{r setup, include=FALSE}
knitr::opts_chunk$set(echo = FALSE, message = FALSE, warning = FALSE)
library(learnr)
library(gradethis)
library(forcats)
library(tidyr)
source("R/setup/gradethis-setup.R")
source("R/setup/tutorial-setup.R")
# Read app parameters
params <- yaml::yaml.load_file("www/app_parameters.yml")
# Benötigte Daten laden
source("R/setup/functions.R")
data_raw <- get_data_raw()
community <- get_community()
plastics_processed <- get_pl_proc()
audit <- get_audit()
wb_areas <- get_wb_areas()
wb_processed <- process_wb_areas(wb_data = wb_areas)
```
```{r results='asis'}
cat(get_license_note(rmarkdown::metadata$title, rmarkdown::metadata$authors))
```
Nachdem wir uns in der vergangenen Woche mit den Grundverben des **dplyr-Packages** die Basics der Datentransformation angeschaut haben, werden wir nun einen Schritt weiter gehen und uns mit den **fortgeschrittenen Methoden der Datentransformation** beschäftigen. Wie gehen wir mit fehlenden Werten um oder fügen ganze Datensätze zusammen - und was versteckt sich eigentlich hinter dem Begriff "Pivoting"? Finden wir's heraus!
---
# **Umgang mit fehlenden Werten (NAs)**
Aus dem `dplyr`-Package kennen wir nun die verschiedenen Funktionen, mit deren Hilfe wir Variablen und Beobachtungen auswählen, filtern oder verändern können. Dabei setzen wir voraus, dass die Daten, die wir transformieren möchten, auch tatsächlich in unserem Datensatz vorhanden sind. Doch was, wenn uns bestimmte Daten fehlen?
## **Schritt 1: Überprüfung**
Zunächst einmal müssen wir natürlich unsere Daten importieren - wir haben das an dieser Stelle bereits für Euch gemacht. Anschließend sollten wir **überprüfen**, ob denn tatsächlich alle Daten vorliegen oder Werte fehlen. Schließlich müssen wir herausfinden, wo Handlungsbedarf ist und wo wir direkt weitermachen können. Dafür checken wir mithilfe der **`sum()`**-Funktion einzelne Werte in unseren beiden Datensätzen, die wir vergangene Woche erstellt haben. <br>
Versucht zuerst einmal, die Summe für die Variable `n_pieces` im Datensatz `community` zu berechnen.
``` {r 08mean_na1, exercise = TRUE}
# Berechnung der Summe
sum(community$n_pieces)
```
Sieht gut aus - im Datensatz `community` sind keine fehlenden Werte enthalten, die bei der Berechnung der Summe stören könnten! Versucht in folgendem Codeblock das Gleiche noch einmal für den `audit`-Datensatz:
``` {r exercise_08mean_na2, exercise = TRUE}
# Hier Euer Code!
```
```{r exercise_08mean_na2-solution}
# Berechnung der Summe
sum(audit$n_pieces)
```
```{r exercise_08mean_na2-check}
grade_this_code()
```
Ups - wir erhalten ein NA. Das steht für "**N**ot **A**vailable" (zu dt. "nicht verfügbar"). Mit diesem Ausdruck gibt uns R das Zeichen, dass **ein (oder mehrere) Wert(e) fehlen** und die gewünschte Berechnung somit nicht ausgeführt werden kann. Wir wissen nun also schon einmal, dass im Datensatz `audit` fehlende Werte für die Variable `n_pieces`vorliegen. Noch wissen wir allerdings nicht, um wie viele fehlende Werte es sich dabei handelt, aber auch das können wir herausfinden - versucht das doch einmal selbst! <br>
*Tipp: Experimentiert mit der Funktion `summary()`!*
``` {r exercise_08mean_na3, exercise = TRUE}
# Hier Euer Code!
```
``` {r exercise_08mean_na3-solution}
# NAs bestimmen
summary(audit)
```
``` {r exercise_08mean_na3-check}
grade_this_code()
```
Sehr gut! Uns wird neben den üblichen Kennzahlen (Minumum, Maximum, Median, etc.) auch die Anzahl der **NAs** angegeben: In diesem Fall betrifft das 11.821 Werte. Klingt erst einmal nach sehr viel, aber trotzdem wollen wir in solchen Fällen durchaus statistische Kennzahlen berechnen. Wie das funktioniert, schauen wir uns jetzt an! <br>
## **Schritt 2: Berücksichtigung**
Denn trotz der fehlenden Werte, können wir in R die statistischen Kennzahlen berechnen. Dafür müssen wir R allerdings explizit sagen, dass die **NAs bei der Berechnung ignoriert** werden sollen. Das funktioniert über das zusätzliche Argument **`na.rm = TRUE`**, das wir einfach an die Funktion anhängen - vielleicht habt Ihr das in den vorherigen Übungen schon einmal gesehen:
``` {r 08mean_na4, exercise = TRUE}
# Berechnung der Summe unter Ausschluss der NAs
sum(audit$n_pieces, na.rm = TRUE)
```
Perfekt - jetzt weiß R, dass die entsprechenden **NAs ignoriert** werden sollen! Neben dieser einen Möglichkeit, gibt es noch viele weitere Wege, NAs in Datensätzen zu entdecken und zu filtern - versucht dafür mal den folgenden Codeblock zu verstehen:
``` {r 08mean_na5, exercise = TRUE}
# Anzahl an fehlenden Werten (is.na gibt für eine Beobachtung TRUE (1) oder FALSE (0) zurück, deshalb können wir das hier aufsummieren)
na_count <- sum(is.na(audit$n_pieces))
# Auflisten von Beobachtungen, die fehlende Werte haben
na_rows <- audit[!complete.cases(audit$n_pieces), ]
na_rows <- which(is.na(audit$n_pieces))
# Neuer Datensatz ohne fehlende Werte
new_df <- na.omit(audit)
```
Für die Variable `n_pieces`in unserem `audit` Datensatz summieren wir also zunächst alle fehlenden Werte und listen die betroffenen Beobachtungen anschließend auf. Auf Basis dieser Transformation können wir dann einen neuen Datensatz ohne die fehlenden Werte erstellen. Die Funktion `**na.omit()**` entfernt dabei alle Zeilen oder Beobachtungen, die fehlende Werte (NA) enthalten, sodass nur vollständige Datensätze erstellt werden.
## **Besonderheiten**
Der Umgang mit fehlenden Werten ist allerdings nicht immer gleich, denn manchmal können diese Werte nicht einfach so ausgeschlossen werden, weil das sonst die **Aussagekraft unserer Analysen** beeinträchtigen würde. Bei [Allison 2010](https://statisticalhorizons.com/wp-content/uploads/Allison_MissingData_Handbook.pdf){target="_blank"} findet Ihr eine gute Übersicht gängiger Herausforderungen und Lösungen. Unser Tipp: Schon bei der Datenerhebung ansetzen und von Beginn an auf genau solche Vorkommnisse prüfen, sodass Ihr rechtzeitig reagieren könnt.
Häufig kommt es zudem vor, dass **fehlende Werte nicht als "NAs" codiert** sind: Wie Ihr vielleicht schon mal gesehen habt, werden fehlende Werte häufig auch als **“N/A”, “N A”, und “Not Available”, oder -99, oder -1** (oder in unserem Fall: "EMPTY") angegeben. Deshalb schauen wir vor der Datenbereinigung und -analyse immer ins zugehörige **Codebuch** des Datensatzes, in dem die Codierung der Variablen erläutert wird. Ansonsten berechnen wir möglicherweise einen Mittelwert, in den fälschlicherweise diese Zahlen einfließen. Aber auch in einem solchen Fall können wir uns wieder auf das `dplyr`-Package verlassen, das eine einfache Lösung parat hat: Die Funktion **`dplyr::na_if()`**. Diese wenden wir mithilfe von **`dplyr::mutate_if()`** auf alle Spalten an (in diesem Fall auf alle numerischen Spalten mithilfe der Bedingung `is.numeric`):
``` {r 08mean_na6, exercise = TRUE}
# Erstellung eines Beispieldataframes mit NAs als "NA" und "-99"
df <- tibble::tribble(
~name, ~x, ~y, ~z,
"Person 1", 1, -99, 6.7,
"Person 2", 3, NA, -99,
"Person 3", NA, 0.76, -1.6
)
# Definition der NAs
df2 <- df %>%
dplyr::mutate_if(is.numeric, dplyr::na_if, -99)
# Erste fünf Zeilen anzeigen
head(df2)
```
Wir sehen, dass alle Werte, die uns zuvor als **-99** ausgegeben wurden, nun ebenfalls als **NAs** codiert sind.
---
# **Pivoting**
## **Was ist Pivoting?**
**Pivoting von Daten** (zu dt. schwenken) ist ein Begriff, der in der Datenanalyse verwendet wird, um eine Umstrukturierung oder Neuorganisation von Daten darzustellen - insbesondere bei Tabellen. Diese Form der Datentransformation ist nicht zwingend notwendig, manchmal aber durchaus nützlich, weil wir Daten somit aus verschiedenen Perspektiven oder Formen betrachten können. Manche Datensätze werden somit durchaus lesbarer, was für die Analyse hilfreich sein kann. Wir unterscheiden zwischen zwei Hauptarten des Pivoting:
1. Pivoting von **breit zu lang** (Long-Format)
2. Pivoting von **lang zu breit** (Wide-Format)
## **Pivoting in RStudio**
Zur Pivotierung unserer Daten können wir zwei Funktionen des `tidyr`-Packages nutzen: `pivot_longer()` und `pivot_wider`. <br>
### **Pivoting: Lang zu Breit (Wide-Format)**
Bei dieser Form des Pivoting werden Zeilen in Spalten umgewandelt, um eine kompaktere Darstellung zu schaffen.
In R nutzen wir dafür die Funktion **`pivot_wider()`**, um Datensätze **breiter zu machen**, d.h. wir erhöen die Anzahl der Zeilen und reduzieren die Anzahl der Spalten. Diese Form der Datenmanipulation ist dann hilfreich, wenn wir eine übersichtliche Zusammenfassung unserer Daten brauchen, z. B. für Präsentationen oder Tabellenkalkulationsprogramme wie Excel.
Beispiel: Wir haben eine Tabelle, in der je eine Spalte Informationen zu unseren Produkten, den Monaten und den entsprechenden Verkaufszahlen entält. Diese Tabelle können wir entsprechend umformen, ohne Informationen zu verlieren:
```{r, echo=FALSE, fig.cap="Graphische Veranschaulichung von `pivot_wider()`", out.width="60%"}
knitr::include_graphics("https://raw.githubusercontent.com/CorrelAid/rlernen_umwelt/main/abbildungen/08_datentransformation/pivot_wider().jpg")
```
Wenn Ihr noch mehr über diese Funktion wissen wollt, könnt Ihr noch einmal in die [R-Dokumentation des tidyverse](https://tidyr.tidyverse.org/articles/pivot.html#wider){target="_blank"} schauen. <br>
### **Pivoting von "Breit" zu "Lang" (Long-Format)**
Bei dieser Form des Pivoting werden Spalten in Zeilen umgewandelt, was durchaus sinnvoll ist, wenn wir mehrere Spalten haben, die die gleichen Informationen für verschiedene Kategorien enthalten.
In R nutzen wir dafür die Funktion **`pivot_longer()`**, um Datensätze zu **verlängern**, d.h. wir reduzieren die Anzahl der Spalten und erhöhen die Anzahl der Zeilen. Diese Form der Datenmanipulation ist dann hilfreich, wenn wir Daten mit verschiedenen Zeitpunkten oder Kategorien haben und diese einfach in Visualisierungen oder Analysen verwenden möchten, z. B. für Zeitreihenanalysen.
Beispiel: Wir haben eine Tabelle, in der jede Spalte für einen Monat steht und die Zeilen die Verkaufszahlen pro Produkt für die jeweiligen Monate enthalten. Durch Pivoting von breit zu lang entsteht eine Tabelle mit den Spalten Produkt, Monat und Verkäufe:
```{r, echo=FALSE, fig.cap="Graphische Veranschaulichung von `pivot_longer()`", out.width="60%"}
knitr::include_graphics("https://raw.githubusercontent.com/CorrelAid/rlernen_umwelt/main/abbildungen/08_datentransformation/pivot_longer().jpg")
```
Mehr zu dieser Funktion findet Ihr ebenfalls in der [R-Dokumentation zum tidyverse](https://tidyr.tidyverse.org/articles/pivot.html#longer){target="_blank"}. <br>
## **Interaktive Übung**
Auch in unserem `audit` Datensatz könnte das Pivoting hilfreich sein: In diesem Datensatz gibt es nämlich eine Reihe kryptischer Variablen, die für die **verschiedenen Plastikarten** stehen, aus denen der gesammelte Müll besteht. Um den Datensatz zu bereinigen, müssen wir diese Variablen so transformieren, dass sie als eine einzige Variable **`plastic_type`** im Datensatz vorkommen:
``` {r 08pivot_longer, exercise = TRUE}
# Ihr nehmt Euren Datensatz...
plastics_processed %>%
#..pivotiert die Plastikarten
tidyr::pivot_longer(
cols = c(hdpe, ldpe, o, pet, pp, ps, pvc), # Betroffene Variablen
names_to = "plastic_type", # Zielspalte
values_to = "n_pieces" # Wertspalte
) %>%
# ..und faktorisiert sicherheitshalber die anderen Variablen (nicht immer notwendig)
dplyr::mutate(dplyr::across( # Zu Faktor konvertieren
.cols = c(country, continent, year, plastic_type),
.fns = as_factor
))
```
Mit `dyplr::pivot_wider()` könnten wir den Datensatz auch wieder in das breite Format bringen, wobei wir Euch empfehlen, die lange Form zu nutzen.
---
# **Datensätze zusammenfügen**
Es kann allerdings auch vorkommen, dass ein einzelner Datensatz uns nicht alle Informationen liefert, die wir für unsere Arbeit brauchen. In einem solchen Fall müssen wir **mehrere Datensätze zusammenfügen** - mit R kein Problem! <br>
## **Interaktive Übung**
Im Exkurs ["Eigene API-Anfragen" zur Session Datenimport](https://correlaid.shinyapps.io/05_1_datenimport-exkurs-api/){target="_blank"} haben wir bereits mit einem Datensatz der Weltbank zu Naturschutzgebieten (`wb_areas`) gearbeitet. In Form von verschidenen Indikatoren, liefert uns diese Datenbank verschiedene umweltbezogene Daten, die für unsere Analyse oder die Arbeit vom #Breakfreefromplastic-Netzwerk interessant sein könnten - z.B. bedrohte Fischarten, Anteil Naturschutzgebiete, usw. Wir werden nun wieder mit diesem Datensatz arbeiten, allerdings ist es auch kein Problem, wenn Ihr den Exkurs (noch) nicht angeschaut habt! Verschafft Euch doch zuerst nochmal einen kleinen Überblick über den Datensatz `wb_areas`, den wir bereits für Euch geladen haben:
``` {r exercise_08gdppc, exercise = TRUE}
# Hier Euer Code!
```
``` {r exercise_08gdppc-solution}
# NAs bestimmen
summary(wb_areas)
```
``` {r exercise_08gdppc-check}
grade_this_code()
```
Die Indikatoren liefern uns verschiedene umweltbezogene Datenn: **ER.PTD.TOTL.ZS** gibt beispielsweise an, wie viel Prozent der Landes- und Wasserfläche aus Naturschutzgebieten besteht.
Den Datensatz `wb_areas` wollen wir nun mit dem `community`-Datensatz zusammenfügen. Damit das gelingen kann, brauchen wir eine **gemeinsame Variable**, die R dafür verwenden kann, um die Länder einander zuordnen zu können. Diese gemeinsame Variable muss in beiden Datensätzen den **gleichen Namen** haben oder von uns zugeordnet werden. Außerdem muss die Kodierung (hier ISO2C) übereinstimmen. Da Ländernamen fehleranfällig sind, fokussieren wir uns auf die **Country Codes**:
``` {r 08wb_clean, exercise = TRUE}
# Umbennung der Spalten
wb_areas %>%
dplyr::select(countrycode = 'iso2c', protected_area = 'ER.PTD.TOTL.ZS')
```
Versucht jetzt einmal herauszufinden, wie viele Länder in beiden Datensätzen `community` und `wb_areas` vorhanden sind (hier führen viele Wege zum Ziel!):
``` {r 08_datenbereinigung, exercise = TRUE}
# Hier Euer Code!
```
``` {r 08_datenbereinigung-solution}
# Länder bestimmen
summary(community$country)
summary(wb_areas$country)
```
``` {r 08_datenbereinigung-check}
grade_this_code()
```
```{r 07quiz_datenbereinigung5}
quiz(caption = NULL,
question("Wie viele Länder sind im Datensatz der World Bank enthalten?",
answer("3"),
answer("266", correct = TRUE),
answer("51"),
answer("4084"),
correct = "Richtig, im Datensatz der World Bank sind insgesamt 266 Länder enthalten!",
incorrect = "Leider nicht ganz richtig, versuche es nochmal!",
allow_retry = TRUE,
try_again_button = "Nochmal versuchen"
)
)
```
```{r 07quiz_datenbereinigung6}
quiz(caption = NULL,
question("Wie viele Länder sind im Datensatz von Breakfreefromplastic enthalten?",
answer("51", correct = TRUE),
answer("4"),
answer("9296"),
answer("4084"),
correct = "Richtig, wir kennen den Datensatz von Breakfreefromplastic bereits. Darin sind insgesamt 51 Länder enthalten!",
incorrect = "Leider nicht ganz richtig, versuche es nochmal!",
allow_retry = TRUE,
try_again_button = "Nochmal versuchen"
)
)
```
Wir wissen nun, wie viele Länder in den jeweiligen Datensätzen vorhanden sind und können jetzt die beiden Datensätze zusammenfügen. Da in dem Datensatz `wb_areas` viel mehr Länder vorkommen als in `comunity`, gibt es verschiedene Möglichkeiten, wie wir beide zusammenfügen können:
- `inner_join`: nur die Länder, die **in beiden Datensätzen** enthalten sind, werden beibehalten
- `full_join`: **alle Länder** aus beiden Datensätzen sind enthalten
- `left_join`/`right_join`: nur die **Länder aus dem zuerst bzw. zuletzt genannten Datensatz** bleiben enthalten
- `left_join()`: Länder aus dem **zuerst** genannten Datensatz bleiben enthalten
- `right_join()`: Länder aus dem **zuletzt** genannten Datensatz bleiben enthalten
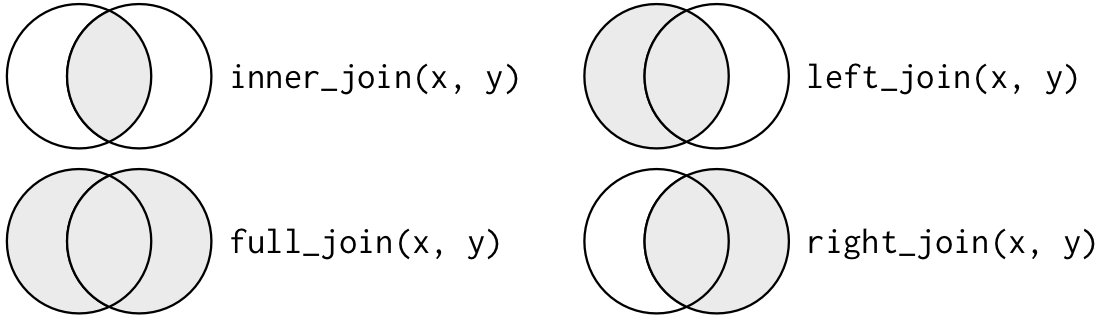{#id .class width=80% height=100%}
Führt die folgenden Codeblöcke nacheinander aus und schaut Euch an, wie diese verschiedenen Joins für unseren Datensatz in der Praxis funktionieren. Was könnt Ihr erkennen? <br>
*Tipp: Um einen kompletten Überblick über den "neuen" Datensatz zu bekommen, müsst Ihr ein bisschen scrollen.*
``` {r 08inner_join, exercise = TRUE}
# Ihr nehmt Euren ersten Datensatz...
community %>%
# ...und führt den Join aus
dplyr::inner_join(wb_processed, by = "countrycode")
```
``` {r 08full_join, exercise = TRUE}
# Ihr nehmt Euren ersten Datensatz...
community %>%
# ...und führt den Join aus
dplyr::full_join(wb_processed, by = "countrycode")
```
``` {r 08left_join, exercise = TRUE}
# Ihr nehmt Euren ersten Datensatz...
community %>%
# ...und führt den Join aus
dplyr::left_join(wb_processed, by = "countrycode")
```
``` {r 08right_join, exercise = TRUE}
# Ihr nehmt Euren ersten Datensatz...
community %>%
# ...und führt den Join aus
dplyr::right_join(wb_processed, by = "countrycode")
```
```{r 08quiz_datenbereinigung8}
quiz(
caption = NULL,
question("Wie Ihr sehen könnt, führt der full_join zu den meisten Zeilen im Datensatz und der inner_join zur geringsten Anzahl an Ländern. Welcher der beiden anderen Joins führt zu einer höheren Anzahl an Ländern im Datensatz?",
answer("left_join"),
answer("right_join", correct = TRUE),
correct = "Gut gemacht! Da wir mit dem Befehl `right_join()` bestimmen, dass die Länder des zuletzt genannten Datensatzes `wb_areas` enthalten bleiben sollen, kommen wir auf insgesamt 266 Länder.",
incorrect = "Das ist leider nicht ganz richtig. `left_join()` bedeutet, dass die Länder des zuerst genannten Datensatzes enthalten bleiben, wohingegen `right_join()` bedeutet, dass die Länder des zuletzt genannten Datensatzes enthalten bleiben. Welcher Datensatz wird hier zuerst bzw. zuletzt genannt?",
allow_retry = TRUE,
try_again_button = "Nochmal versuchen"
),
question("Welche der folgenden Aussagen sind wahr?",
answer("Beim Inner Join bleiben nur Zeilen bestehen, die in beiden Datensätzen vorhanden sind (n = 50)", correct = TRUE),
answer ("Beim Full Join bleiben alle Zeilen bestehen, passende werden zusammengefügt (n = 267)", correct = TRUE),
answer ("Beim Left Join bleiben alle Zeilen des links referenzierten Datensatzes (community) bestehen, passende werden zusammengefügt (n = 51)", correct = TRUE),
answer ("Beim Right Join bleiben alle Zeilen des rechts referenzierten Datensatzes (wb_areas) bestehen, passende werden zusammengefügt (n = 266)", correct = TRUE),
correct = "Gut gemacht!",
incorrect = "Leider nicht ganz richtig, versuche es nochmal! Wenn Du dir unsicher bist, kannst Du oben noch einmal die verschiedenen Möglichkeiten des Joinings nachlesen.",
allow_retry = TRUE,
try_again_button = "Nochmal versuchen"
)
)
```
---
# **Und jetzt Ihr!**
Nun ist es wieder an der Zeit, das Gelernte praktisch anzuwenden. Versucht, die **R Datei: 08_datentransformation-uebung.R** im [Übungsordner](https://download-directory.github.io/?url=https://github.com/CorrelAid/rlernen_umwelt/tree/main/uebungen){target="_blank"} unter "08_datentransformation" zum Laufen zu bringen. Darin sind noch einmal ein paar Übungen zum Pivoting und Joining von Datensätzen, mit denen Ihr alles noch einmal ausprobieren könnt. Versucht diese Übungen nachzuvollziehen und zu bearbeiten. Wie immer: Kein Problem, wenn nicht alles auf Anhieb klappt - bringt alle offenen Fragen gerne mit in die nächste Live-Session!
---
# **Zusätzliche Ressourcen**
- [Schummelblatt: tidyr](https://github.com/CorrelAid/rlernen_umwelt/blob/main/cheatsheets/06_cheatsheet-tidyr.pdf){target="_blank"} (engl.)
- [Data Cleaning in R](https://app.dataquest.io/course/r-data-cleaning){target="_blank"} auf DataQuest (engl.)
- [Advanced Data Cleaning in R](https://app.dataquest.io/course/r-data-cleaning-advanced){target="_blank"} auf DataQuest (engl.)
<a class="btn btn-primary btn-back-to-main" href=`r params$links$end_session`>Session beenden</a>