-
Notifications
You must be signed in to change notification settings - Fork 0
/
Copy pathGWAS.qmd
34 lines (18 loc) · 1.88 KB
/
GWAS.qmd
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
---
title: "GWAS"
editor: visual
---
## ¿Qué es un GWAS?
Genome Wide Association Studies (GWAS) son análisis en los que se estudia la asociación entre variantes a lo largo del genoma y un fenotipo. De manera sencilla, se buscan variantes que estén más presentes en individuos con cierto fenotipo. El fenotipo estudiado puede ser un rasgo continuo o la presencia o ausencia de algo, por ejemplo si se tiene o no un enfermedad.
## ¿Qué es un summary statistic?
Estas estadísticas resumidas continene la información del análisis estadístico para cada variante. Esto es el tamaño del efecto que tiene ese variante, la desviación estandar, un p-valor asociado entre otros valores.
## ¿Qué es el tamaño del efecto?
La fuerza de asociación entre la variante y el fenotipo de interés.
El tipo de análisis estadístico usado para medir la fuerza de asociación depende del fenotipo, para fenotipos cuantitativos usamos la regresión lineal, mientras para fenotipos binarios usamos la regresión logística.
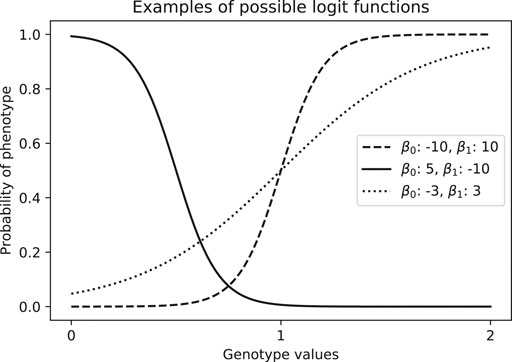
Figura tomada de [Jaiswal et al., 2019](https://www.nature.com/articles/s41598-019-41602-6)
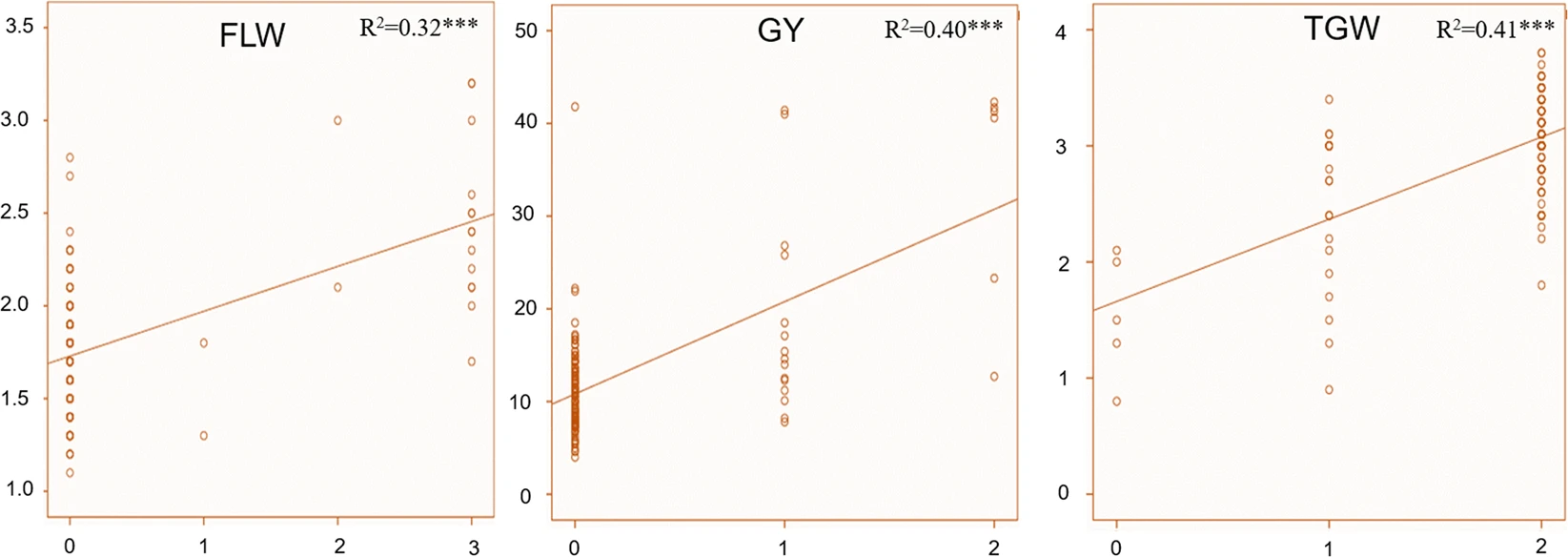
Figura tomada de [Robles-Espinoza et al., 2021](https://www.frontiersin.org/journals/genetics/articles/10.3389/fgene.2021.703901/full)
Si quieren más información les recomiendo leer este artículo: [An Overview of Strategies for Detecting Genotype-Phenotype Associations Across Ancestrally Diverse Populations](https://www.frontiersin.org/journals/genetics/articles/10.3389/fgene.2021.703901/full)
## ¿Con qué software lo podemos hacer?
Por ejemplo [Regenie](https://rgcgithub.github.io/regenie/)