-
Notifications
You must be signed in to change notification settings - Fork 1
Applications_ru
Основным направлением использования открытой библиотеки для извлечения признаков из метагеномных данных является анализ данных секвенирования микробиоты кишечника человека при различных состояниях и сопутствующих заболеваниях. Поиск маркеров, связанных со сложно диагностируемыми заболеваниями, является актуальной задачей. Поскольку микробиота кишечника тесно связана со всеми системами организма и влияет на иммунный ответ, разумным предположением является наличие в ней составляющих, которые могут появляться как ранний сигнал или, наоборот, в результате различных заболеваний.
Библиотека была применена для поиска признаков в наборах данных пациентов с воспалительными заболеваниями кишечника. Пациенты были разбиты на три когорты: здоровые, с язвенным колитом и с болезнью Крона. Были извлечены признаки и обучены предсказательные модели, которые могут быть использованы в качестве вспомогательного метода для поддержки принятия врачебных решений при диагностировании заболеваний.
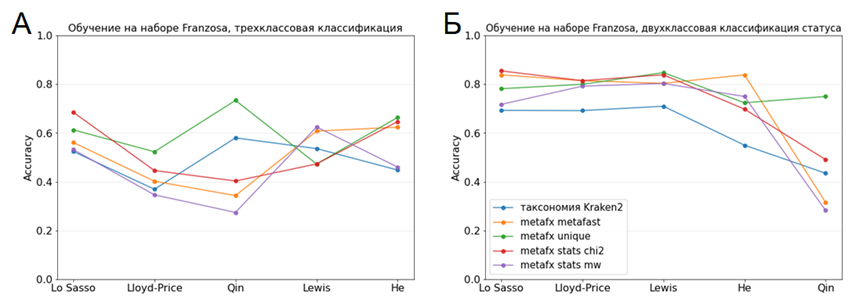
Из открытых источников были отобраны и использовались шесть наборов данных метагеномного секвенирования кишечника пациентов, страдающих воспалительными заболеваниями кишечника (ВЗК): Franzosa, Lo Sasso, Lloyd-Price, He, Qin, Lewis.
К каждому из выбранных обучающих наборов были применены четыре алгоритма для извлечения признаков. На полученных матрицах признаков обучена логистическая регрессия и выполнено предсказание классов образцов из всех остальных наборов данных, посчитана метрика MCC (Matthew Correlation Coefficient) и метрика точности (accuracy).
Точность бинарной классификации статуса пациента (болезнь Крона – язвенный колит) значительно превышает качество трехклассовой классификации и достигает значения 0.85 по метрике accuracy при обучении на наборе Franzosa и предсказании набора Lo Sasso. При предсказании других наборов методом metafx unique значение метрики accuracy не опускается ниже 0.75. Это существенно превосходит по качеству результаты моделей, обученных на данных таксономической аннотации, и позволяет увеличить точность классификации в среднем на 10 %. Результаты классификации по метрике accuracy представлены на рисунке ниже.
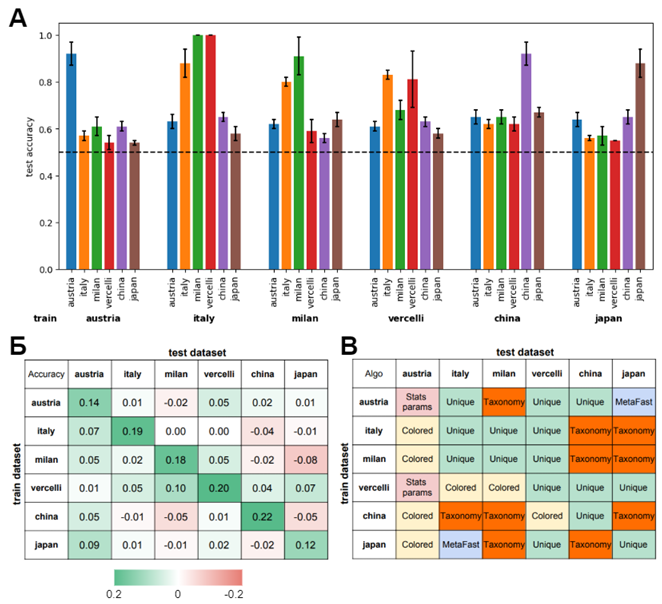
Для анализа были выбраны данные секвенирования микробиоты кишечника людей, связанные с раковыми заболеваниями. Было принято решение использовать для анализа три категории пациентов: больные колоректальным раком (CRC), больные колоректальной аденомой (adenoma), контрольная группа – условно здоровые пациенты (control). Использовались наборы данных из четырех исследований: Austria, Italy, China, Japan.
Для извлечения признаков из каждого набора данных были использованы четыре алгоритма из разработанной библиотеки MetaFX. Алгоритм metafx metafast был применен как базовый алгоритм извлечения признаков без учителя в качестве наиболее простого метода, не использующего информацию по группировке метагеномных образцов на категории. Алгоритмы metafx colored и metafx unique были использованы как методы извлечения признаков с использованием информации о категориях образцов. Кроме того, был использован алгоритм metafx stats в двух модификациях: с параметрами по умолчанию и с ручным подбором оптимальных параметров для каждого набора данных.
Результаты работы моделей логистической регрессии в задаче трехклассовой классификации представлены на рисунке ниже. А) Точность лучшего алгоритма для пары обучающий набор (группировка, выделен шрифтом внизу) и тестовый набор (столбцы, разные цвета); Б) Разница точности между лучшим алгоритмом MetaFX и алгоритмом на основе таксономических признаков; В) Выбранный лучший алгоритм для каждой пары обучающий-тестовый набор.